先进成像技术能够对复杂生命现象实现多维多尺度的观察,但随之产生的大量数据却给图像分析带来了巨大挑战,如何准确高效地分析海量成像数据已经成为揭示生命科学新机制的关键一环。
2022年7月12日,清华大学脑与认知科学研究院戴琼海团队受Nature Methods邀请,以Challenges and opportunities in bioimage analysis为题发表技术评论【1】,总结生物图像分析的新进展和新趋势,并展望新兴技术所带来的新的可能性。

“More is different (更多就是不同)【2】。”正如菲利普··安德森大约50年前所指出的那样,大规模基本单元的相互作用可能会催生新的属性,而这些新属性无法用基本单元的简单规律来解释。生命科学是一门表现出鲜明层级结构的学科,其范围涵盖从基因、蛋白质、细胞器、细胞到组织、器官和个体。先进测量仪器能够实现不同尺度下生物结构和动态的观测,但在连接细胞和器官乃至个体的介观尺度成像方面存在长期的不足。为了填补这一空白,荧光显微镜的数据通量在过去十年中增加了好几个数量级,为研究各种生理病理状态下大规模细胞间的相互作用开辟了新途径。为了适应成像仪器不断攀升的数据通量,如何准确高效地处理、分析和理解大规模成像数据以催化生命科学新发现正变得越来越重要。
新进展和新趋势
大约十年前,深度卷积神经网络首次展示了其在计算机视觉领域的统治地位。从那时起,深度学习因其在性能和效率方面的关键优势而成为当代图像分析的支柱。这场革命迅速延伸到显微成像领域,科学家针对不同的生物应用和成像模式提出了各种各样的智能化方法,以解决图像增强、分类、分割、细胞追踪等各种问题。长期以来,监督学习一直是并且目前仍然是基于深度学习的图像分析的主要范式,如果有足够多的训练图像以及与之配对的真值(标签),那么针对特定图像分析任务构建具有良好性能的模型并不困难。然而,在显微成像领域很难甚至有时完全不可能获得配对的真值,对真值的依赖已经成为监督学习在生物图像分析中不可避免的本质缺陷。近年来,生物图像分析领域最令人印象深刻的趋势是从传统的监督学习向自监督和无监督学习的转变(图1)。使用这些新的训练机制,网络可以自主地学会完成特定任务,而无需任何配对的真值进行网络的训练【3-5】。此外,显微成像的一个典型特征是不同模式生物、实验条件和实验室之间的数据之间存在很大差异,预训练模型往往会产生伪影甚至完全无法应用于新数据,自监督和无监督学习则提供了更好的解决方案来为特定数据训练定制的模型。对于大规模图像分析,因为原始数据本身就是大规模的训练集,因此自监督和无监督方法具有先天的优势,这将为训练具有更好泛化能力的大型模型铺平道路。
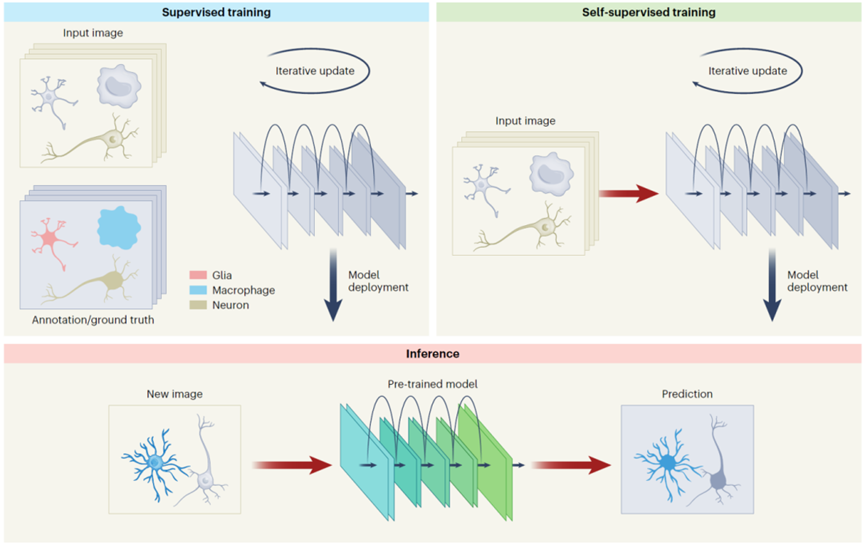 图 1:用于生物图像分析的监督学习、自监督/无监督学习方法。
图 1:用于生物图像分析的监督学习、自监督/无监督学习方法。
不仅是学习机制,深度神经网络的架构也在不断推陈出新以超越经典的卷积网络。Vision Transformer是一种使用自注意力机制来提取内在特征的新型网络架构,在各种计算机视觉任务上几乎都能带来显著的性能提升。它们捕获长程依赖的能力可以克服卷积核的局限性,从而更好地集成全局信息。正如Transformer在从长 DNA 序列预测遗传变异方面所取得的成功一样【6】,挖掘大规模成像数据中的远程时空相关性来发现新的生物学机制也具有巨大的潜力。例如,在神经功能成像中,Transformer可以帮助揭示长时程记录中两个遥远事件之间的因果关系,以及在介观尺度成像中表征两个遥远神经元之间的关联。
作为一种植根于人类和其他动物决策过程的计算模型,强化学习被广泛用于构建智能体,通过奖励正确的行为并惩罚错误的行为来学习与环境的交互。与图像分析相结合,强化学习有望揭示大规模成像数据中隐藏的模式。例如,将迁移细胞视为智能体,将周围的其他细胞建模为环境,深度强化学习可以推断胚胎发育过程中细胞迁移的机制【7】。强化学习也能够用于破译动物行为的神经机制,如果能够通过实验获得大量的神经成像数据以及同步的感官刺激,就可以使用强化学习以数据驱动的方式构建认知模型。这种范式可以扩展到不同的领域,通过筛选不同器官不同生理病理状态下的大规模成像数据来获取新的生物学发现。深度强化学习的另一个成功是训练智能体完成各种具有挑战性的任务,例如玩视频游戏、下棋等等【8】。这给显微成像带来了启发,通过合理地设计奖励并迭代地从大量实验数据中学习,显微镜可以学会与样本交互,从而以最佳参数完成给定的成像任务,并自动发现有趣的现象。
成像是一个严格的光学过程,需要足够可靠才能支撑科学发现。作为一种定量测量手段,生物图像中每个像素的强度都具有特定的生物物理和生物化学意义,例如钙成像中的钙离子浓度、电压成像中的膜电位、空间转录组中的基因表达等。因此,在图像处理之后依然保留这种定量特性对于解码潜在的生物现象至关重要。对于旨在提高图像质量的低级视觉任务(如去噪、超分辨率重建、去模糊等),将图像形成的物理原理纳入处理框架可以提高结果的可信度。在计算成像领域,更好地对成像过程进行建模将带来更好的成像性能【9】,这已经是一个经过广泛验证的事实。
面临的挑战
尽管近年来生物图像分析取得了令人印象深刻的成就,但仍然存在一些问题阻碍了这些工具在显微成像和生命科学研究中的广泛使用。文章总结了几个要点,希望研究界通过共同的努力来解决这些问题。
标准和指标。发布标准验证数据集并设置相应的性能指标可以规范生物图像分析方法的发展。由于不同成像方式的图像差异很大,因此应为每种成像方式存档特定的数据集。这些验证数据集必须具有足够的代表性,并涵盖足够广泛的样本和成像条件。为了更直观地对各种方法进行对比和排名,每个数据集的性能指标最好是能够反映综合性能的单个数字。此外,分析效率务必要被包含在性能指标中,以便于在大规模图像分析中的实际应用。
可解释性和可靠性。尽管能够取得优越的性能,深度学习却因其黑盒属性而在显微成像中遭遇信任危机。赋予深度神经网络可解释性是一个长期存在的挑战,这可能需要最底层理论的创新。但一些技术手段如特征可视化和基于物理的建模可以在一定程度上提高可靠性。大多数研究人员最担心的是结果中可能存在潜在的伪影,如果能够开发切实可用的方法来定量评估结果的可靠性,例如在输出结果的同时提供相应的置信水平,将有助于缓解深度学习的信任危机。
通用平台。最先进的图像分析方法建立在计算机视觉的最新进展之上,使用这些方法需要强大的编程技能和专业背景,这对于没有相关专业背景的生物学家来说往往比较困难。最成功的生物图像分析平台是基于 ImageJ的Fiji,而目前迫切需要一个更新的Fiji,或者一个新的交互式平台来推动智能生物图像分析的广泛应用。一个好的平台应当不仅能够部署预训练模型,更重要的是能够训练新的模型,这些操作都依赖强大的算力,因此平台必须考虑如何方便地获取本地或云端的计算资源。此外,由于目前的深度学习方法大多基于Python,因此新平台应兼容Python,以充分利用开源资源。
数据共享。生物成像巨大的数据量使得数据共享变得相当困难,为了促进大规模数据集的共享,首先需要一个支持高维成像数据的在线预览和下载的网络平台,其次采用高效无损的压缩方法可以降低对传输带宽和存储设备的要求。此外,开发一种将原始大规模数据集划分为若干独立可用单元的新的数据格式将是十分有用的,这样每个单元都可以被独立地预览、下载和使用。这种“元数据”不仅应包含数据集的基本描述,还应包含数据缩略图, 用户无需下载整个数据集即可通过元数据对数据的形式和内容有具体的了解。
新兴技术提供了新的可能性
人工智能的最新研究表明,大语言模型(Large Language Models)可以在语言理解、推理和编程等方面具有人类水平的表现,其中ChatGPT和多模态的GPT-4因其能够理解用户并流畅地回答各种问题而引起了广泛关注。大模型所表现出的巨大潜力为构建生物图像分析的专用大模型提供了新的可能,这就像训练一位能够理解需求并自动处理成像数据的人工智能工程师,可以为研究人员编写自定义脚本甚至是训练特定的深度学习模型。用户只需上传数据、在对话框中发送具体的需求并给出一些中间指示,大模型就能完成图像分析任务(图2),这将有效减轻大规模图像分析的沉重负担。然而,要积累足够多的数据并训练出如此智能和专业的模型还有很长的路要走,公开已发表论文的代码和数据对于实现这一长期目标至关重要。另外值得注意的一点就是,目前的大语言模型有时会给出虚假的答案,必须制定严格的标准和验证手段,才能确保它们在图像分析中发挥恰当的作用。
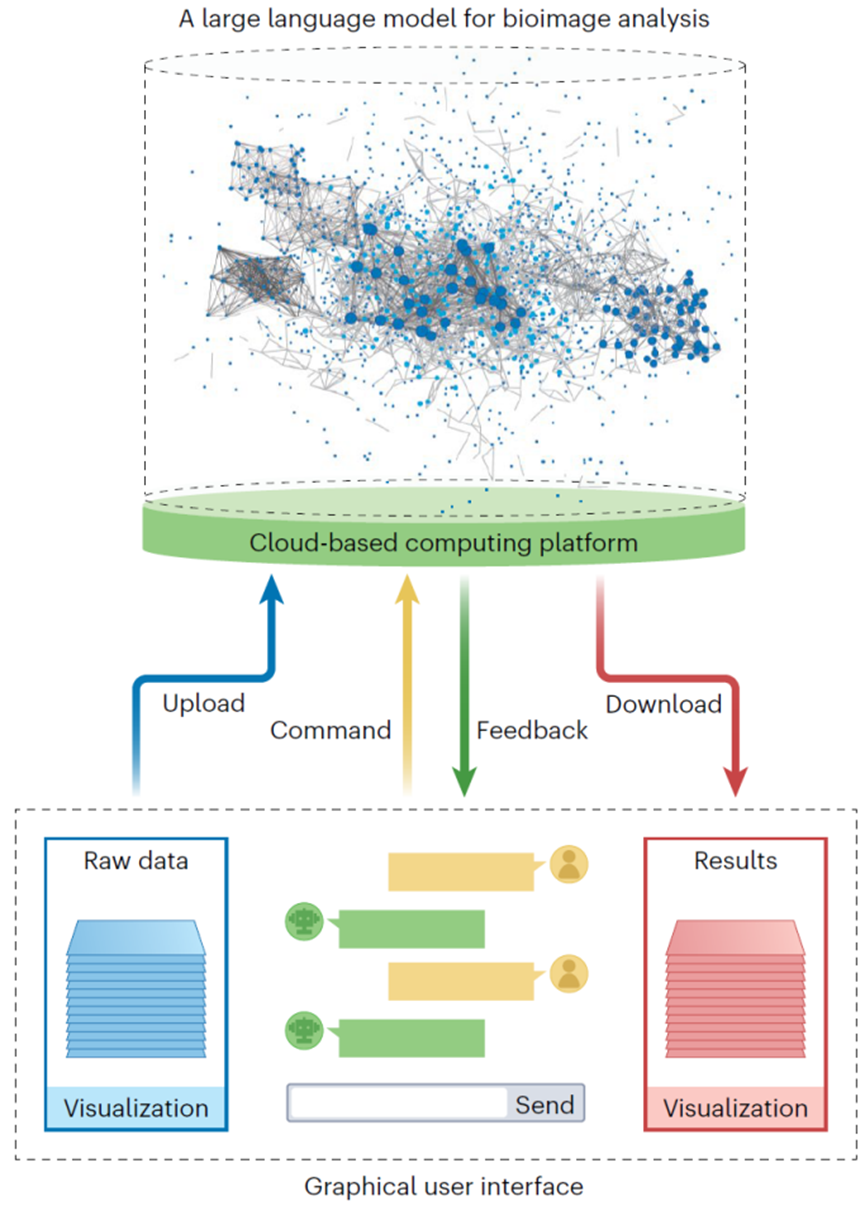 图2 :用于生物图像分析的人工智能大模型
图2 :用于生物图像分析的人工智能大模型
大规模图像分析日益增长的需求同样对计算能力提出了艰巨的挑战,传统的硅基处理器很难实现大规模数据的实时处理。光计算是一种新兴技术,它利用光子代替电子以光速处理信息,从而大幅提升数据的速度【10】。集成化的光子环路不仅可以实现深度神经网络的推理,还可以用于实现基本的矩阵运算。将图像分析与光计算相结合,有望将处理速度提升到更高的水平,并实现更多大规模、高通量的生命科学和生物医学应用。
清华大学自动化系助理研究员李欣阳、张元龙为该文章共同第一作者;清华大学脑与认知科学研究院、清华大学自动化系戴琼海教授、吴嘉敏助理教授为该论文共同通讯作者。
原文链接:https://www.nature.com/articles/s41592-023-01900-4
参考文献
[1] X. Li, Y. Zhang, J. Wu, and Q. Dai, "Challenges and opportunities in bioimage analysis," Nature Methods, vol. 20, no. 7, pp. 958-961, 2023, doi: 10.1038/s41592-023-01900-4.
[2] P. W. Anderson, "More is different: broken symmetry and the nature of the hierarchical structure of science," Science, vol. 177, no. 4047, pp. 393-396, 1972.
[3] X. Li et al., "Reinforcing neuron extraction and spike inference in calcium imaging using deep self-supervised denoising," Nat Methods, vol. 18, no. 11, pp. 1395-1400, Nov 2021, doi: 10.1038/s41592-021-01225-0.
[4] B. Midtvedt et al., "Single-shot self-supervised object detection in microscopy," Nat Commun, vol. 13, no. 1, p. 7492, Dec 5 2022, doi: 10.1038/s41467-022-35004-y.
[5] X. Li et al., "Unsupervised content-preserving transformation for optical microscopy," Light-Sci Appl, vol. 10, no. 1, p. 44, Mar 1 2021, doi: 10.1038/s41377-021-00484-y.
[6] Z. Avsec et al., "Effective gene expression prediction from sequence by integrating long-range interactions," Nat Methods, vol. 18, no. 10, pp. 1196-1203, Oct 2021, doi: 10.1038/s41592-021-01252-x.
[7] Z. Wang, Y. Xu, D. Wang, J. Yang, and Z. Bao, "Hierarchical deep reinforcement learning reveals novel mechanism of cell movement," Nat Mach Intell, vol. 4, no. 1, pp. 73-83, Jan 2022, doi: 10.1038/s42256-021-00431-x.
[8] V. Mnih et al., "Human-level control through deep reinforcement learning," Nature, vol. 518, no. 7540, pp. 529-33, Feb 26 2015, doi: 10.1038/nature14236.
[9] J. Wu et al., "An integrated imaging sensor for aberration-corrected 3D photography," Nature, vol. 612, no. 7938, pp. 62-71, Dec 2022, doi: 10.1038/s41586-022-05306-8.
[10] X. Lin et al., "All-optical machine learning using diffractive deep neural networks," (in English), Science, vol. 361, no. 6406, pp. 1004-1008, Sep 7 2018, doi: 10.1126/science.aat8084.